人工智慧賦能原位結構生物學 提出冷凍電鏡顆粒挑選新方法
中國網/中國發展門戶網訊 近日,中國科學院自動化研究所多模態人工智慧系統實驗室楊戈團隊與中國科學院生物物理研究所蛋白質科學研究平臺生物成像中心孫飛團隊合作,以人工智慧技術賦能原位結構生物學,提出了一種基於弱監督深度學習的快速準確顆粒挑選方法DeepETPicker,相關研究以《DeepETPicker:基於弱監督深度學習的快速準確三維冷凍電子斷層掃描圖像粒子自動檢測器》(DeepETPicker: Fast and accurate 3D particle picking for cryo-electron tomography using weakly supervised deep learning)為題發表于《自然·通訊》(Nature Communications)。
生物大分子(如蛋白質)的結構與功能會隨著細胞生理狀態的變化不斷進行動態調整。原位結構生物學是在接近自然生理狀態下研究生物大分子結構和功能的科學,而原位冷凍電鏡技術(cryo-electron tomography, Cryo-ET)以其高解析度和在接近生理條件下觀察樣品的特點,成為原位結構生物學研究中的關鍵手段。原位冷凍電鏡的技術流程涉及樣品製備、數據採集、電子斷層重建、顆粒挑選、粒子平均等多個步驟。生物大分子的顆粒挑選,即定位識別,是其中一個關鍵環節。受限于Cryo-ET圖像的極低信噪比和重建偽影等因素,成千上萬個目標顆粒的手動挑選極為耗時費力,現有自動挑選方法的應用受到人工標注量高、計算成本高和顆粒品質不理想等多方面限制。
DeepETPicker僅需要少量人工標注顆粒進行訓練即可實現快速準確三維顆粒自動挑選。為降低對人工標注量的需求,DeepETPicker優選簡化標簽來替代真實標簽,並採用了更高效的模型架構、更豐富的數據增強技術和重疊分區策略來提升小訓練集時模型的性能;為提高顆粒定位的速度,DeepETPicker採用圖形處理器(GPU)加速的平均池化-非極大值抑制(MP-NMS, mean pooling and non-maximum suppression)後處理操作,與現有的聚類後處理方法相比提升挑選速度數十倍。此外,為方便用戶使用,項目團隊推出了操作簡潔、界面友好的開源軟體(圖1)以輔助用戶完成圖像預處理、顆粒標注、模型訓練與推理等操作。
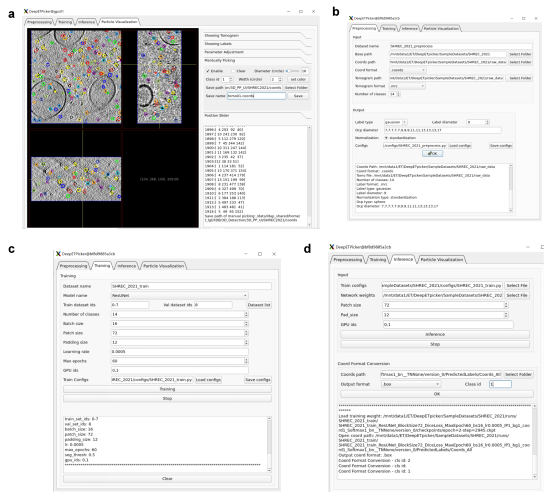
圖1 DeepETPicker 軟體用戶圖形界面
使用DeepETPicker從冷凍電子斷層掃描圖像中挑選顆粒的整體工作流程如圖2所示,包括訓練階段(圖2a-c)和推理階段(圖2e-g)。在訓練數據的準備階段,研究人員優選了弱標簽TBall-M來代替真實掩模以減輕人工標注負擔,並在模型架構設計方面,引入坐標卷積(coordinated convolution)和圖像金字塔(image pyramid inputs)到3D-ResUNet的分割架構中以提高定位的準確性。在模型推理階段,DeepETPicker採用重疊斷層圖分區策略(OT, overlap-tile),避免了由於邊緣體素分割精度不佳而産生的負面影響,進而結合MP-NMS操作加速了顆粒中心定位過程。
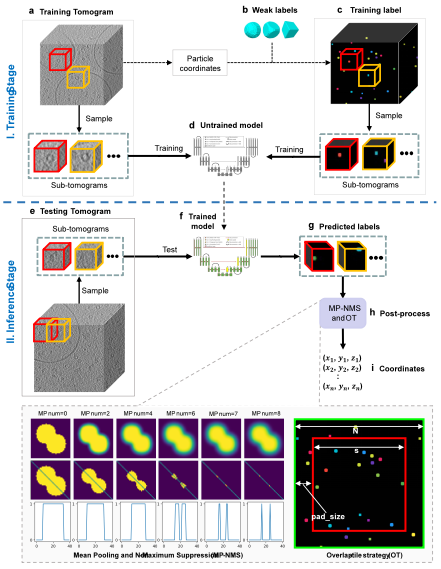
圖2 使用 DeepETPicker 從冷凍電子斷層掃描圖像中挑選顆粒的整體工作流程
研究團隊將DeepETPicker與目前性能最優的顆粒挑選方法在多種冷凍電子斷層掃描數據集上進行了性能評估對比,採用六個定量指標全面評價顆粒挑選的品質(圖3):精確率-召回率(Precision-Recall)、F1-分數(F1-score)、對數似然概率貢獻度(Log-likelihood Contribution)、最大值概率(maximum value probability)、RH解析度(Rosenthal-Henderson resolution)、全局解析度。結果表明,DeepETPicker在倣真與真實數據集上均可實現快速準確的顆粒挑選,其綜合性能明顯優於現有的其他方法,生物大分子結構重建達到的解析度也達到採用專家人工挑選顆粒進行結構重建同樣的水準,這進一步體現了DeepETPicker在原位高解析度結構解析中的實用價值。DeepETPicker將為採用原位冷凍電鏡技術的原位結構生物學研究提供有力的支援。
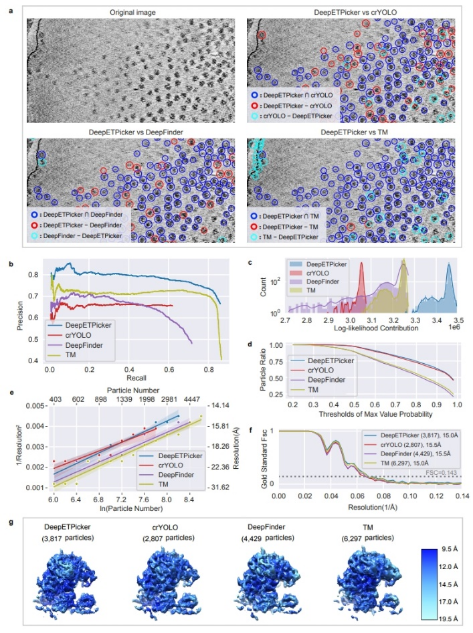
圖3採用定量指標評估DeepETPicker在EMPIAR-10045實驗數據集上的粒子挑選性能
本論文第一作者為中國科學院自動化研究所助理研究員劉國樂和中國科學院生物物理研究所工程師牛彤欣,中國科學院自動化研究所多模態人工智慧系統全國重點實驗室楊戈研究員與中國科學院生物物理研究所生物大分子國家重點實驗室孫飛研究員為該論文的共同通訊作者,楊戈課題組博士生裘夢軒、孫飛課題組研究員朱赟也參與了此項研究。













